论文阅读系列–CNN网络架构篇
1. scGNN is a novel graph neural network framework for single-cell RNA-Seq analyses**
一句话摘要: 利用GNN实现单细胞之间的关系分析并实现聚类。
论文之我问:
1.1 这个论文的动机是什么
众所周知,scRNA-seq分析是十分具有挑战的,先前的研究采用了简单KNN进行分析但并不足以分析复杂的细胞关系,目前GNN作为一种更强有力的工具,通过重现input从而实现更高效的的表征关系。
1.2 这篇论文的研究框架是什么样的
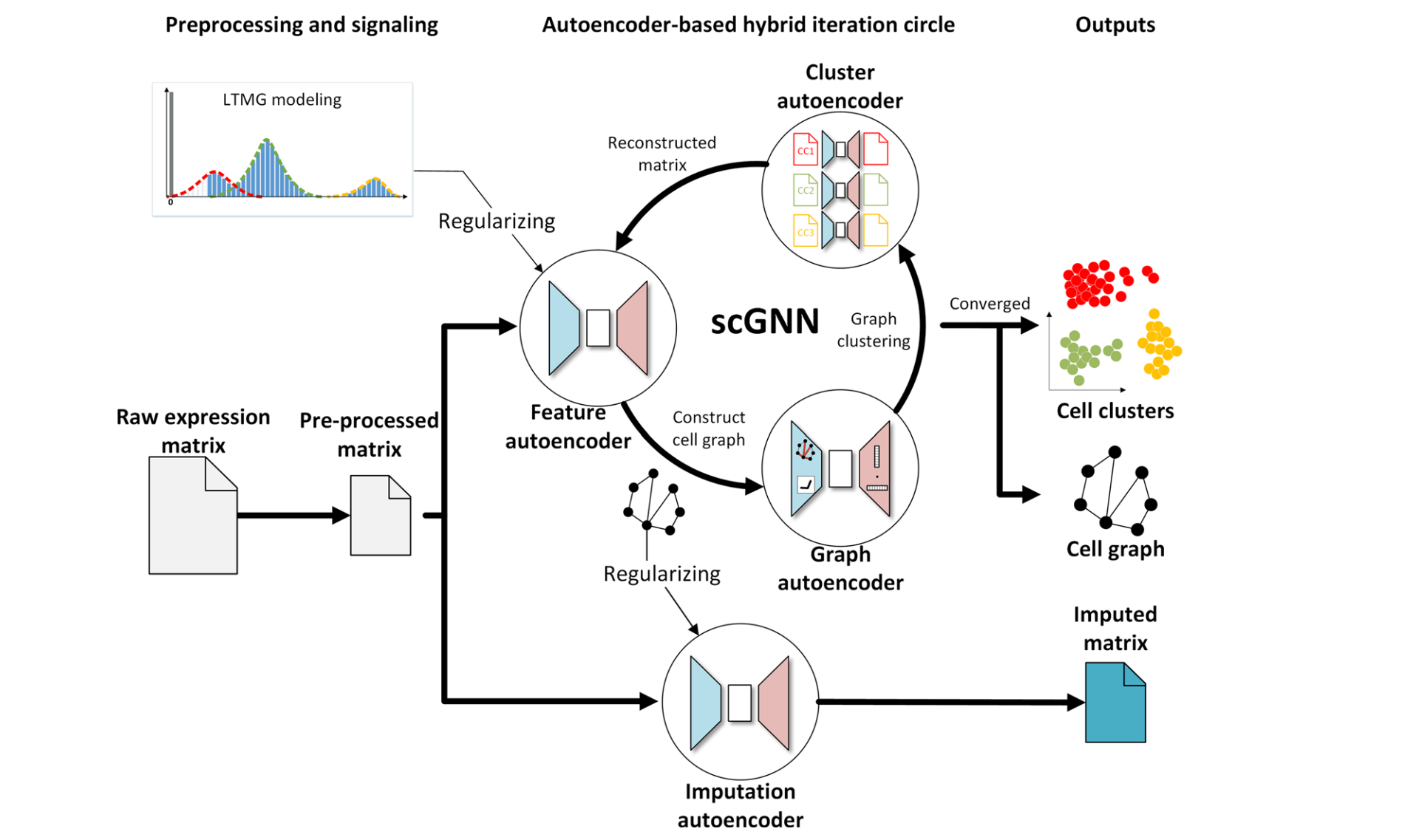
如图1所示。 输入是一个基因表达矩阵,会连续过3个autoencoder然后再循环的过程中完成聚类和图的构建。
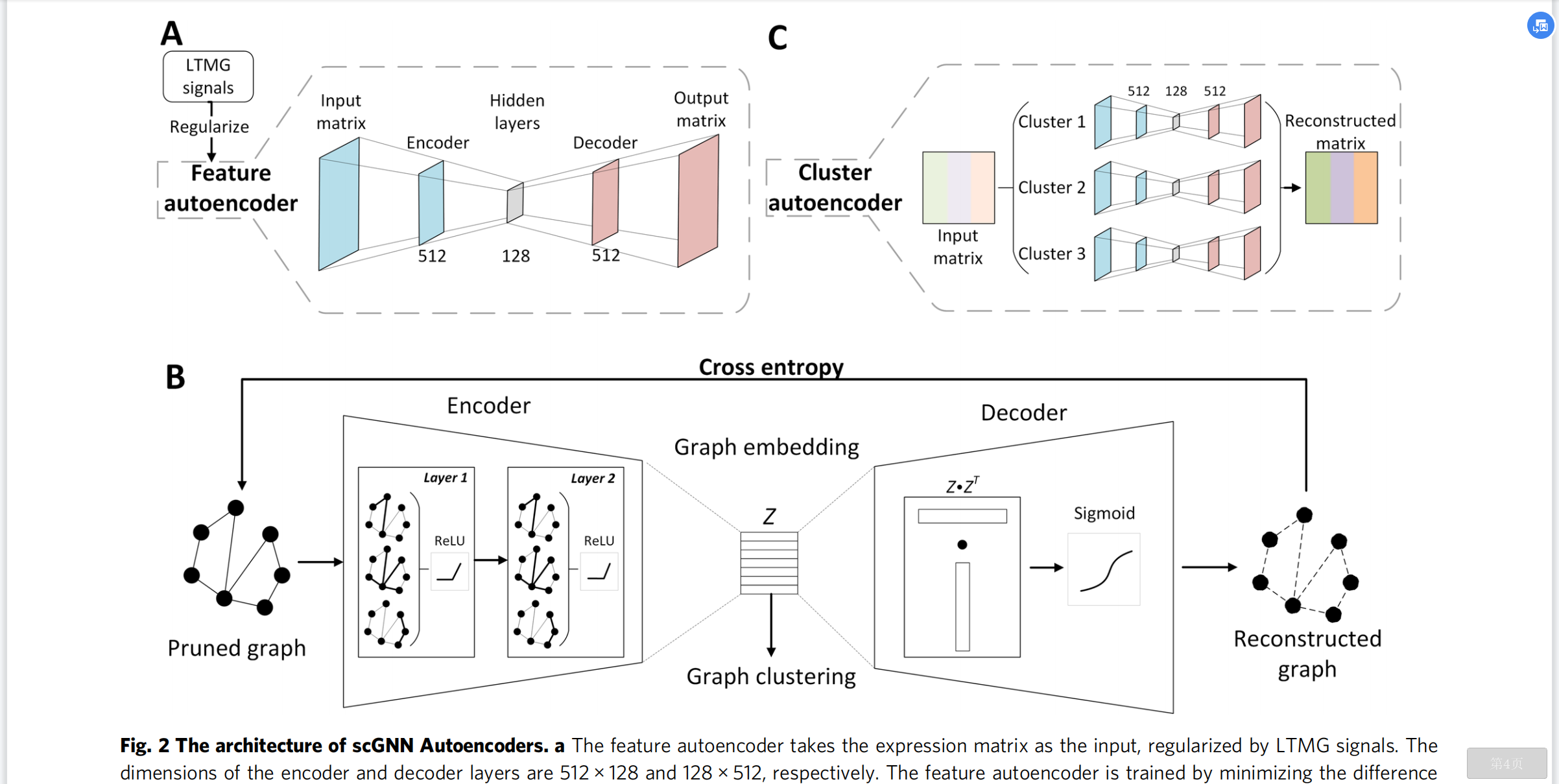
如图2所示。scGNN 自编码器架构。
值得一提的是,B种的输入Graph是由knn构建,然后图的聚类是由k-means and Louvain实现。converge的两个条件分别是(1)图的变化比率小于某个阈值,即图已经稳定,(2)细胞类型的变化比率小于某个阈值,即细胞聚类已经稳定。
1.3 这篇论文的实验效果
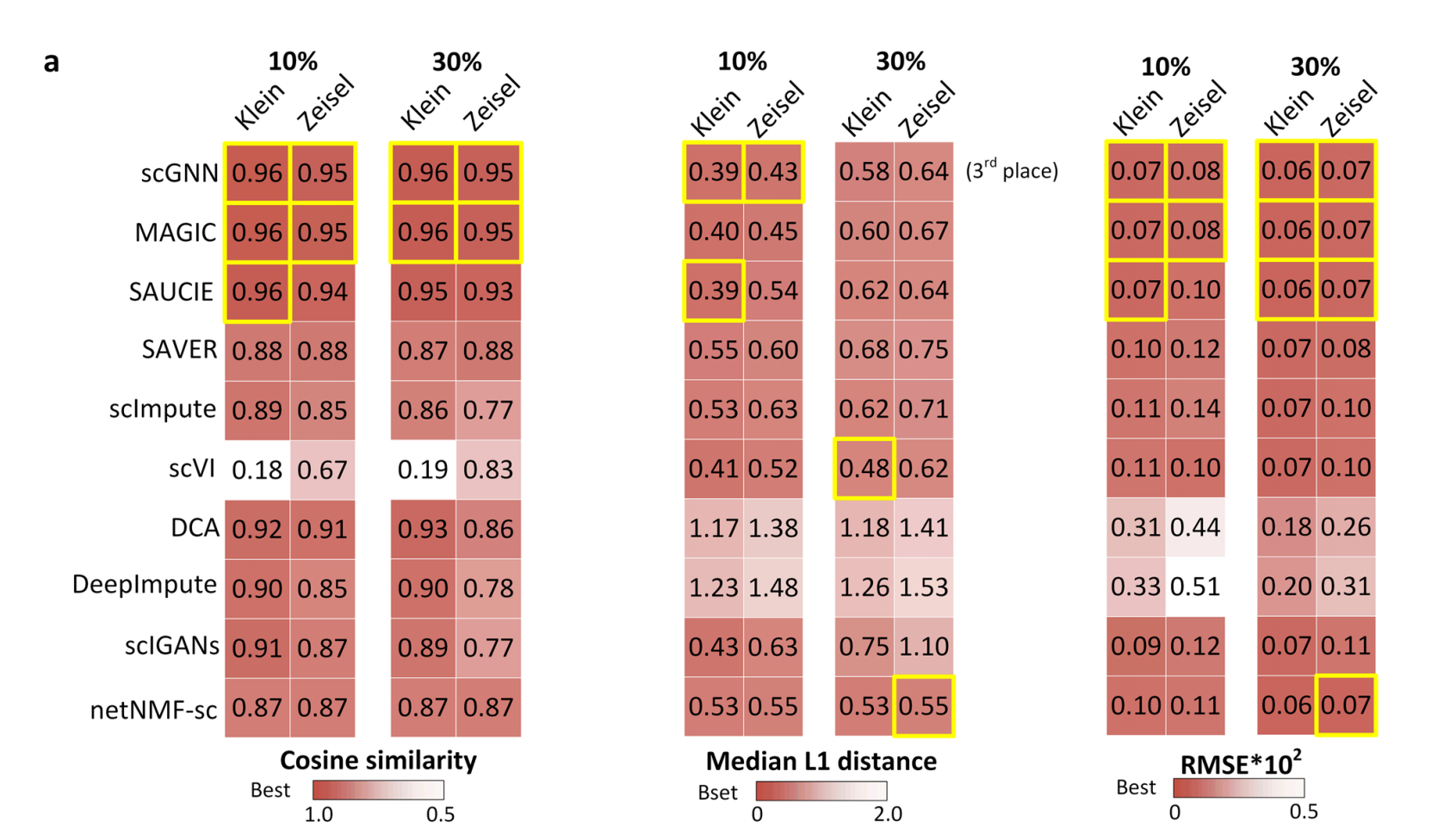
scGNN的一些evaluation, 具体可以参考原文。
